|
|
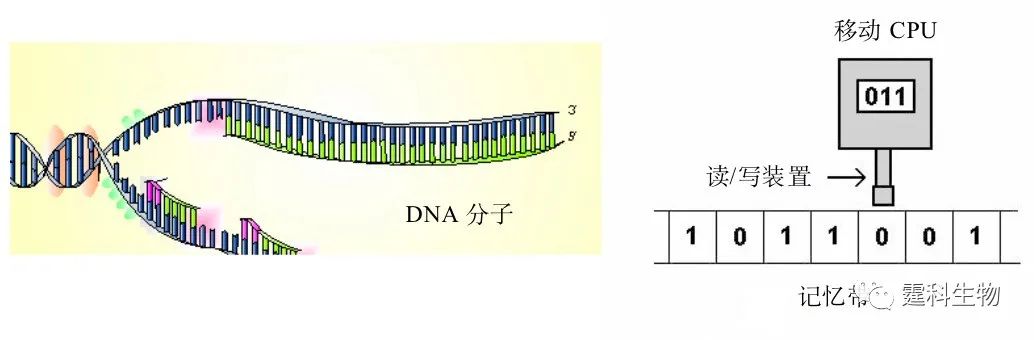
图解微流控|第32期发表时间:2022-01-25 14:25 微流控芯片实验室在核酸研究的应用(四) 10.5 DNA计算机 10.5.1 DNA计算和DNA计算机 DNA计算是一种模拟生物分子DNA的结构并借助于分子生物技术进行计算的新方法。DNA计算机则是使用DNA分子储存信息并利用DNA生化反应进行计算的新型计算机(图10-29)。
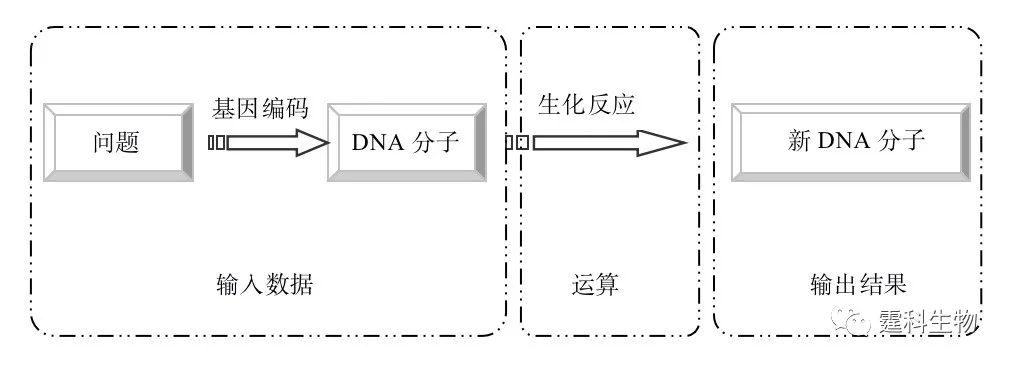
图10-29 以DNA为介质的新型计算机 DNA是生物体中的遗传物质,是由A、G、C、T四种碱基按一定顺序排列聚合成的螺旋状双链大分子。类比电子计算机中0和1编码的字符串,可将DNA序列看成是由A、G、C、T四种符号编码的蕴含一定信息的字符串 它的基本原理是以DNA分子中的基因编码作为存储数据,利用DNA分子在某种酶的作用下瞬间完成某种生物化学反应(杂交、变性、酶连和酶切等)来完成计算,DNA分子在此过程中从一种基因编码变为另一种基因编码。如果将反应前的基因编码作为输入数据,那么反应后的基因编码就可以作为运算结果,继而完成各种不同的运算过程[31]。 DNA计算的基本原理如图10-30所示。
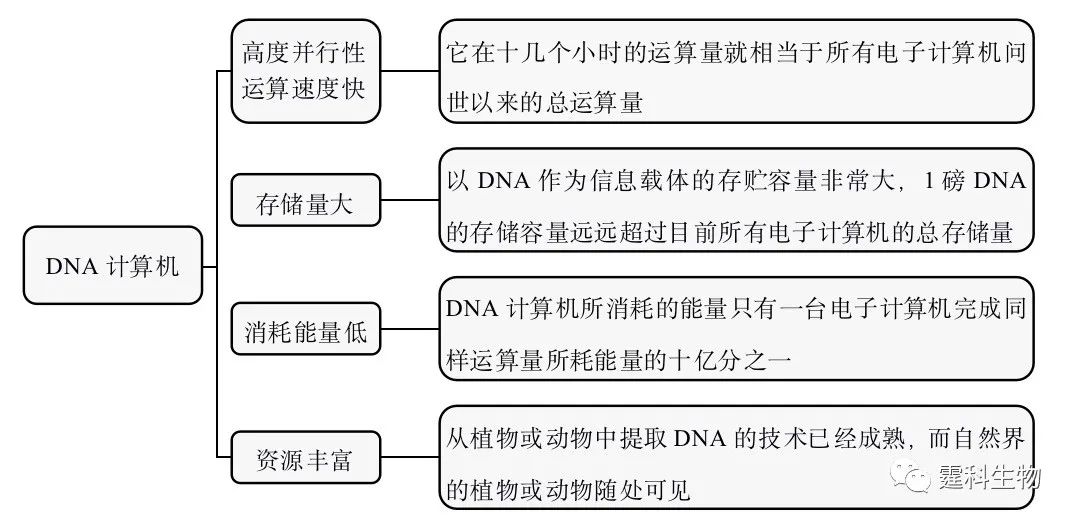
图10-30 DNA计算的基本原理 DNA计算通过对DNA链进行丰富的精确可控的生化反应,包括标记、扩增或者对原有链的破坏来完成各种不同的运算过程,以DNA作为介质的新型的计算机具有四个显著优点(图10-31)。
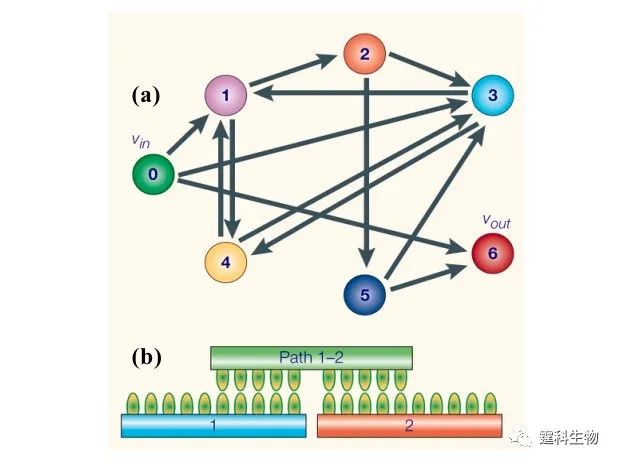
图10-31 DNA计算机的优点 由于DNA计算的上述四大优点,它极大地吸引了不同学科、不同领域,尤其是计算机、生物、数学、物理、化学以及信息领域的科学家的关注。 当然,DNA计算也有它的局限性。首先,这种计算还存在精度问题。与DNA分子相关的生物化学反应,比如PCR和杂交过程都可能出现碱基错配,或者,由于动力学和热力学原因,进行得不彻底或发生副反应,因此对此后的计算产生不良影响,甚至导致结果有误。其次,读出结果的技术瓶颈还有待解决。目前,很多情况下,读出结果所消耗的时间远比DNA计算过程要长,而输出结果也面临类似的问题。因此,整体而言,DNA计算机尚处于研究阶段,离真正的应用还有相当的距离。尽管如此,研究者对DNA计算的前景仍然充满信心,正努力在理论和技术两个层面使之不断完善。 DNA计算机的高度并行性特点,非常适合解决数学中的NP问题,一个成功的案例是利用DNA计算做多项式乘法求解Hamilton问题[31](图10-32)。经典的Hamilton路径问题是给定一组具有m条路径连接的n个城市,要求从给定的城市出发,经过每个城市一次且只有一次并到达给定的终点。Adleman合成了一些DNA片段来代表城市,并合成了连接两个DNA片段的DNA链来代表路径,这些DNA片段在T4 DNA连接酶的作用下连接到一起,形成了所有可能的组合,大量的可能性问题在DNA酶反应瞬间得到高度并行性的快速计算。
图10-32 DNA计算机解决Hamilton路径问题 图10-33所示为利用DNA计算解决可满足性问题(SAT问题)。利用简单的分子生物学操作进行DNA计算,为一个有20个变量、100万种可能结果的数学难题(3-SAT问题)找到了答案,这是迄今利用非电子化计算手段解决的最复杂数学问题[32]。
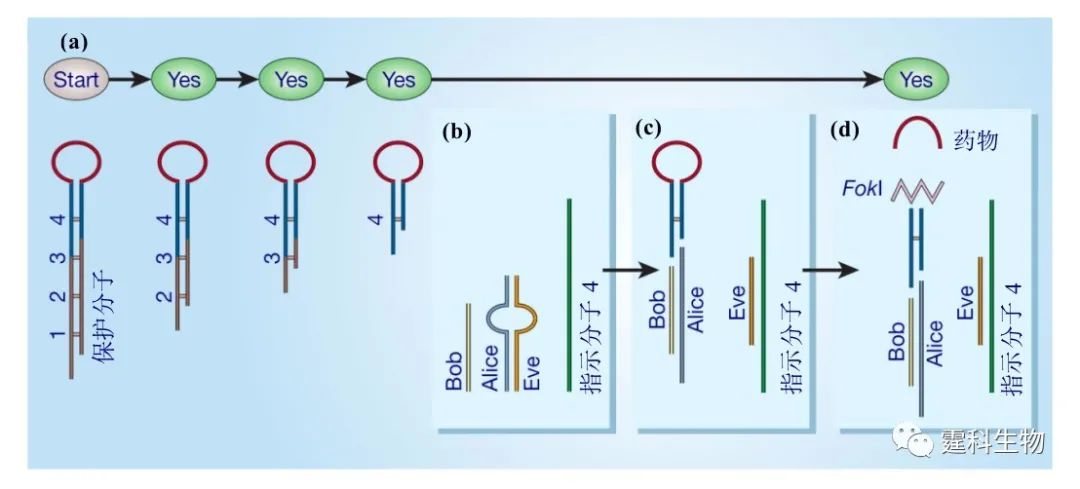
图10-33 解决3-SAT问题所应用的DNA计算机示意图[32] 除解决数学问题外,由DNA和酶构成的可程序化自动机还可被应用于医学诊断和治疗。利用由DNA分子和相应的酶分子构成的DNA计算机,是一部输入和输出、软件和硬件均由生物分子组成的有图灵机功能的可程序自动机[33](图10-34)。
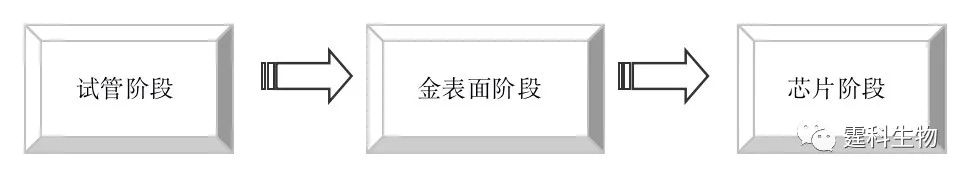
图10-34 DNA分子诊断可程序自动机 自动机的初始状态是一个茎环状结构,末端的“环”是单链的药物分子,“茎”是双链的保护分子,在双链的“茎”的保护下,单链的“环”状药物处于无活性状态。保护分子代表4种符号,与四种基因一一对应(图10-34(a)),当第一种基因表达符合疾病的诊断时(癌基因升高或抑癌基因降低),表示yes的双链指示分子与保护分子的粘性末端相连,由限制性内切酶(FokⅠ酶)切除掉保护分子的第一段,自动机向yes状态转化,否则,自动机将向no状态转化。最后一个诊断状态需要一些特殊结构的DNA分子(图10-34(b))。为实现最终状态的改变,“Eve”分子与指示分子4结合,“Alice”与“Bob”结合后与保护分子4结合(图10-34(c))。当所有转化均为yes时,即FokⅠ酶(锯齿线)识别到“Alice-Bob”,双链的“茎”才会在适当的位置被切断,使得“环”被释放,形成有活性的药物(图10-34(d))。 迄今为止,DNA计算赖以实现的载体主要有三类,即试管、金表面和芯片(图10-35)。微流控芯片技术所具有不同操作单元灵活组合、大规模集成的特点[34],为取代试管或表面操作,构建一个严格意义上的DNA计算机提供了一种理想的平台。近年来已有包括作者实验室在内的个别研究小组展开了这方面的研究。
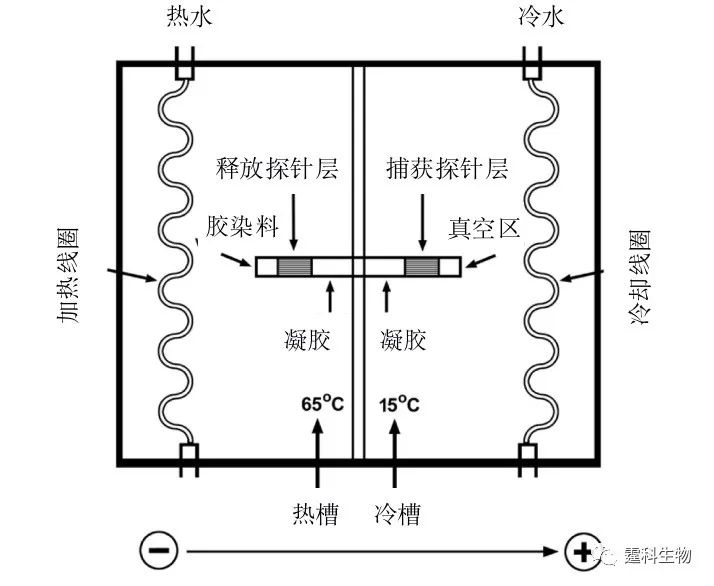
图10-35 DNA计算机发展方向 10.5.2 微流控芯片DNA计算机 微流控芯片作为一个多功能的技术平台非常适合DNA计算研究(图10-36)。DNA计算所涉及的各种生化反应和分离过程如杂交、酶切、酶连、PCR、电泳等均可在微流控芯片上实现。微流控芯片有望成为DNA计算机走向实用化的主流平台。
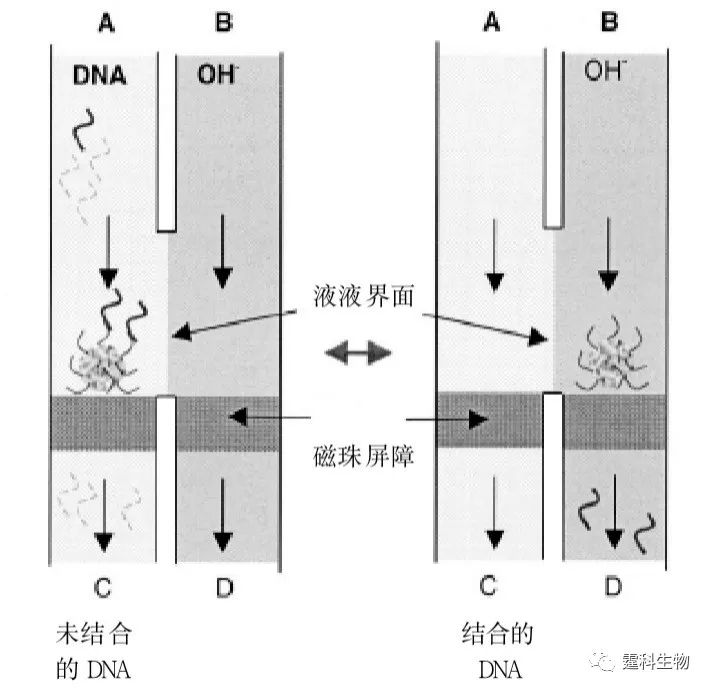
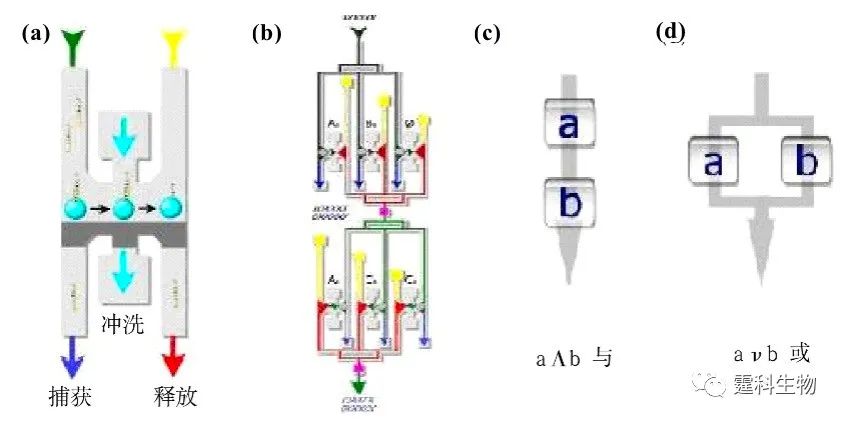
图10-36 以微流控芯片为平台的DNA计算机 (a) 微流控芯片具有微通道、微阀、微泵和微传感器形成的整个系统网络,可以集成控制系统和数字/模拟转换装置等操作单元,各单元相互独立又彼此连通;(b) 在微流控芯片上可实现对反应体系的实时监测,更准确地把握DNA计算的整个过程 利用微流控技术可进行分子计算[35],微流控芯片用作DNA计算的选择性转移模块[36]。芯片通道中包含连有一定DNA链的磁珠,代表备选条件。磁珠在通道A捕获溶液中满足条件的DNA分子,并将其带到通道B,释放出来,流向通道D;未被捕获的DNA分子流向通道C,实现选择性转移(图10-37)。磁珠的转移由外部磁场控制。
图10-37 选择性转移模块示意图[36] 利用图10-38所示微流控芯片DNA计算选择性模块解决含有6个顶点的最大圈问题,建立解决含有20个顶点最大圈问题的微流控芯片模型[37, 38]。
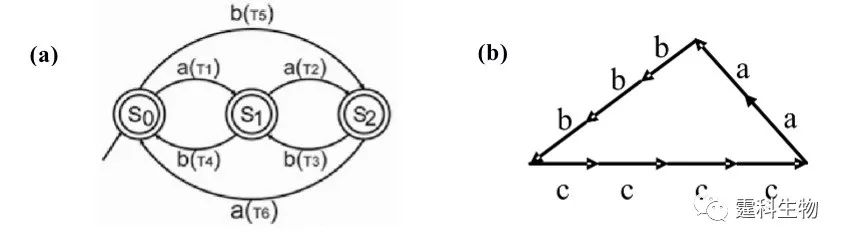
图10-38 利用选择性转移模块实现逻辑控制 (a) 在选择性转移模块的捕获和释放通道间增加了冲洗通道,以去除非特异性吸附的DNA分子。(b)、(c)、(d) 是通过这些模块的串联并联,实现“与”“或”逻辑控制 作者所在课题组和上海交通大学的同事们合作,从2004年开始了基于微流控芯片的DNA计算机的研究工作,并已取得一定进展[39-42]。提出了基于微流控芯片平台的有限状态自动机的基本构想,将计算机的基本功能如输入、输出、计算、控制等单元微流控芯片上完成,用以解决等腰三角形识别问题(图10-39),同时用该模型证实了生物存储器的可行性和必要性。
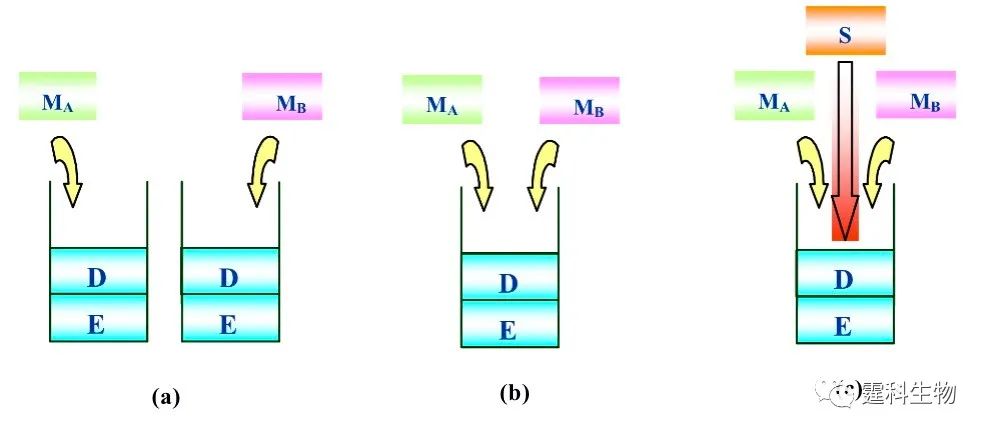
图10-39 用于三角形识别的有限状态自动机模型[39] (a) 比较三角形边长的2-符号3-状态模型,S0为初始状态,S1和S2为最终状态;(b) 三角形被视为由a、b、c基本单元组成的句法结构 在模式识别时,可将三角形看成由一些小线段组成,每个线段有相同的长度。这些线段分成水平线、上行斜线和下行斜线等三种类型,是构成三角形的基本单元,分别用a,b,c表示。在此基础上可把三角形描述成由基元组成的字符串,如图所示的三角形可以表示为“aabbbcccc”。为简便计,对三角形三边进行两两比较。分别用含有a,b的字符串表示三角形的两边,通过比较a,b的个数,进行两边长度的比较。默认S0为初始状态,如果最终状态为S1或S2,则a,b的个数一定不等。由于含3种状态的自动机中存在循环,最终状态为S0时,a,b的个数可能相等或相差3的倍数。这样仅从最终状态不能完全比较a,b的个数。为区分这两种情况,作者设计了生物存储器,记录计算的中间过程。最初建立含两个栈的存储模式。把a,b对应的存储单元分子MA,MB分别存入两个栈,通过比较两个栈中存储分子的长度,就可以比较a,b的个数。但这种存储模式无法存储a,b交叉的字符串,为此要建立含一个栈的存储模式。把a,b对应的存储单元分子MA,MB存入同一个栈,可存储任意字符串。前面的计算中默认S0为初始状态,实际上初始状态可以为S0,S1或S2中的任意一个,这样就需要把含有初始状态信息的片段S也存入栈中。存储模式从简单到复杂,不断完善起来(图10-40)。DNA计算和存储所涉及的各种生化反应均在微流控芯片上得以实现。
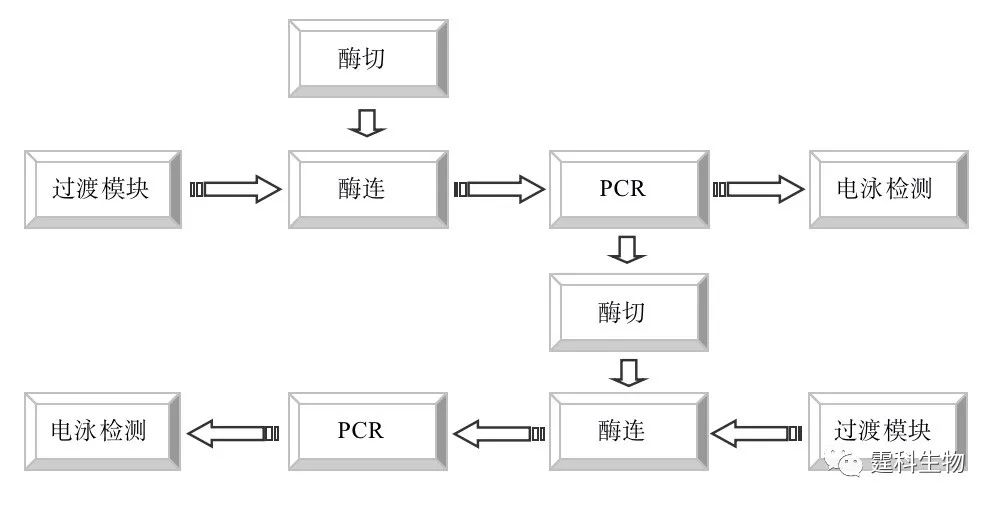
图10-40 DNA计算机多种生物存储模式 存储模式包括(a) 两栈存储、(b) 一栈存储、(c) 状态片段存储。MA、MB为与a、b字符串相对应的存储单元分子,在栈中存储相应的存储单元分子,记录不同的计算最终状态 作者所在课题组已将DNA计算所涉及的技术单元如酶切、酶连、杂交和PCR等置于微流控芯片上完成。所构建的DNA计算机工作流程如图10-41所示,微流控芯片上的微泵、微阀和电极控制等单元技术也被应用于DNA计算单元的衔接,并通过芯片电泳对计算结果进行实时,快速,准确的检测。据此在微流控芯片上完成了计算机的基本功能,为下一步把微流控芯片DNA计算机用于疾病诊断和药物筛选奠定了基础[41]。
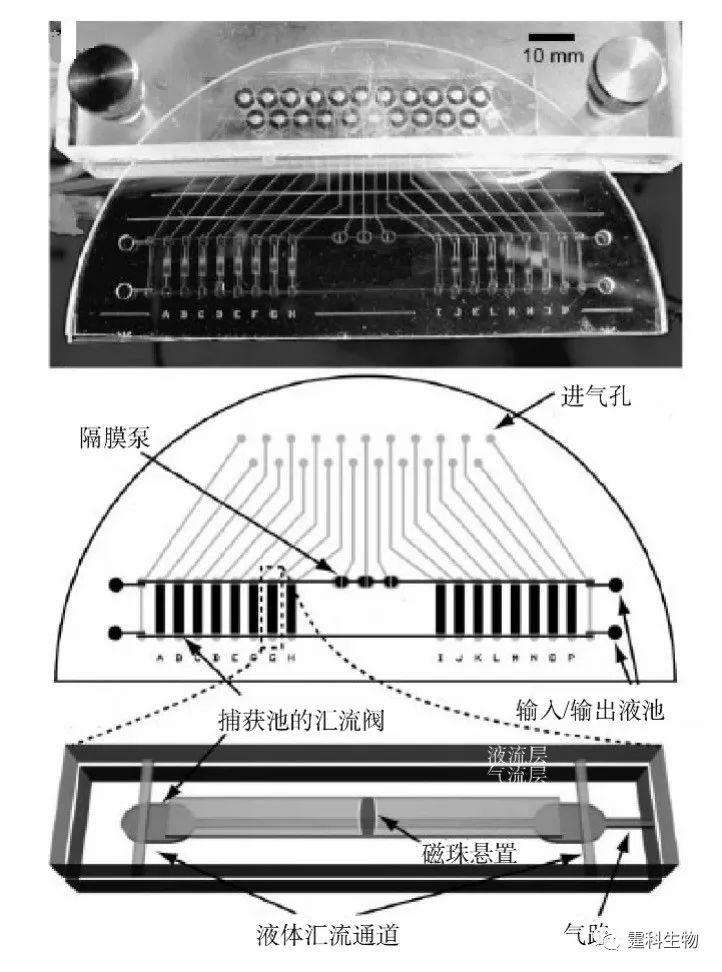
图10-41 DNA计算机工作流程 近期,作者所在课题组构建了一台以微流控芯片为计算载体的DNA计算机模型样机,设计了一种DNA计算方法和三种与乳腺癌原癌基因和抑癌基因相对应的计算分子。在该模型样机上模拟DNA计算机进入机体,对乳腺癌基因表达情况进行计算(诊断),根据计算结果在众多正常细胞中找出少量的肿瘤细胞,自动对识别确认的肿瘤细胞释放自杀基因药物(治疗),对识别确认的正常细胞不显示药物释放功能,因此对其生理功能不产生影响。 图10-42所示为在微流控芯片平台上求解满足性问题[43]。利用单核苷酸多态性以碱基A或T代表二进制数。被编码的DNA单链在微阀和微泵的驱动下依次流经16个捕获池。池中的磁珠连接有代表不同备选条件的DNA单链,能捕获符合条件的DNA单链。通过一系列捕获/冲洗/释放步骤,最终解决了一个3-变量,4-语句的满足性问题。
图10-42 解决满足性问题的微流控芯片实物及示意图[43] 从以上的研究成果可以看出,微流控芯片不同操作单元灵活组合、大规模集成和可控性强的特点使其更适合于DNA计算机的研究工作。它将为今后进行更大规模,更复杂的DNA计算提供有力的技术支撑,有望成为DNA计算机走向实用化的主流平台。 参考文献  文章来源:《图解微流控芯片实验室》林炳承、秦建华 著 为进一步推广普及微流控芯片技术,霆科生物获得林炳承先生授权,公司网站及公众号开辟“图解微流控”专版,连载《图解微流控芯片实验室》一书的主要内容。 本版刊登内容仅限于学术交流,严禁用于任何商业用途。 欢迎转发分享,如需转载请与我们联系,谢谢! |